原始 DNA 数据是一切探索的起点

* 重要提醒:消费级基因检测是价格低廉的个人 DNA 数据探索方式,并不具备临床诊断的功能。使用科研级基因芯片获得一份高质量的 DNA 原始数据,并随研究探索不断获得最新的研究结论,是消费级基因检测最重要的价值。
▽
怎样获得我的 DNA 原始数据?
dna@gesedna.com
中国邮箱发送( 不要使用outlook, yahoo,hotmail,gmail,iCloud 等邮箱,会被拒收)
下单收件人姓名+ 联系电话
你的用户名(不是昵称)+ 个人主页截图
邮件内容里备注「570 万」
▽
了解你的 570万 DNA 原始数据
GSA 芯片是各色使用的第一款高通量芯片。
2018 年 10 月之后,各色全面升级 ASA 芯片联合定制版。这是 Illumina 公司第一次专为亚洲人设计和研发的芯片,也是国内第一款基于 ASA 定制芯片的消费级基因检测产品。
2024 年 5 月之后,各色使用 CGA 芯片,为用户提供服务。CGA 芯片,是在 GSA 芯片基础上,针对中国人群群体结构(包括一些主要少数民族)做了专门优化,更适配中国人群的检测。
在约 70 万原始检测数据的基础上,我们研发团队使用隐马尔可夫(Hidden Markov Model, HMM) 模型,利用全基因组数据作为参考数据集,针对各色用户的测序数据进行了扩展,明确了模型参数与每个用户的每个位点准确率相关关系。
最终,我们使用高性价比和准确率高达 98% 的方式,获得了最能代表人与人差异的 570 万位点。
这就意味着,不需要花钱就能多测 500 万位点。今后,我们会依据这570 万位点,给大家带来更丰富的检测性状和解读。
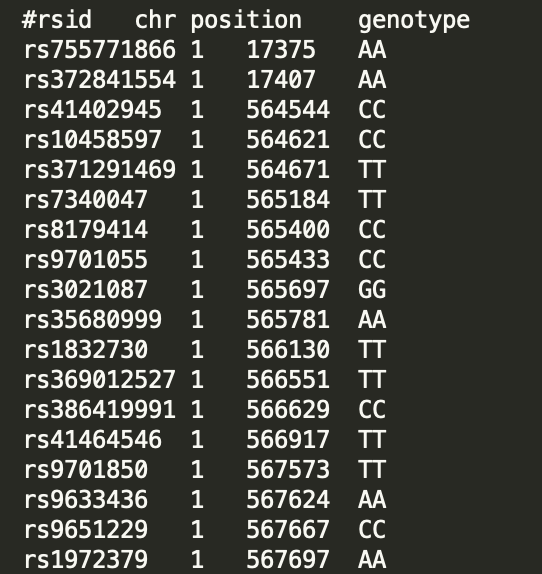
打开你的 DNA 数据,有四列。第一列是基因位点的名称,无实际意义;第二列是染色体,1-22 对应 22 条染色体,X 、Y 代表 X、Y 染色体,MT 代表线粒体 DNA ;第三列是这个基因位点在染色体上的位置;第四列是你的数据类型。主要有三种形式,碱基对:A\T\C\G 四个字母的组合;结构变异,插入和删除:I(insertion)\D(deletion);以及此处位点没有检出:–。
▽
我还有问题
祝你探索愉快:)
